DNA-RNA三链体是双链DNA与单链RNA相互作用形成的异源三链杂交体,在DNA-RNA三链体形成过程中,单链RNA与富含嘌呤的双链DNA通过碱基互补配对,利用Hoogsteen氢键形成并维持三链体的稳定性[1]。相较于传统的沃森-克里克碱基配对,Hoogsteen氢键更加灵活,因此RNA可以更广泛、更灵活地与基因组DNA结合,从而为分子之间的动态相互作用提供了更高的可塑性[2]。近年来,研究发现哺乳动物基因组中含有大量潜在的DNA-RNA三链体,这些三链体广泛分布于注释基因、启动子以及基因间区域[3, 4],但确切功能尚不完全清楚。因此,鉴定功能性DNA-RNA三链体对于深入理解生命过程具有重要意义,也为疾病的诊断和治疗提供新视角。
最近研究表明,DNA-RNA三链体广泛参与基因表达调控、DNA损伤修复、染色体稳定维护和肿瘤发生发展等多个重要的生理和病理过程[5]。然而,DNA-RNA三链体在肿瘤发生发展中的作用机制尚不完全清楚。特别是长非编码RNA(Long noncoding RNAs, lncRNAs)参与组成的DNA-lncRNA三链体在肿瘤发生发展中的关键作用和调控机制仍有待进一步研究。因此,阐明DNA-lncRNA三链体如何形成又如何调控肿瘤发生发展具有十分重要科学意义和潜在临床价值。
2024年10月21日,同济大学生命科学与技术学院张赫教授团队在《自然·通讯》(Nature Communications)上发表了题为“Peptidylprolyl isomerase A guides SENP5/GAU1 DNA-lncRNA triplex generation for driving tumorigenesis”的最新研究成果,率先揭示了肽基脯氨酰异构酶A(PPIA)指导的DNA-lncRNA三链体“招募促癌”机制,首次提出了异构酶辅助DNA-RNA杂交体形成的创新概念。

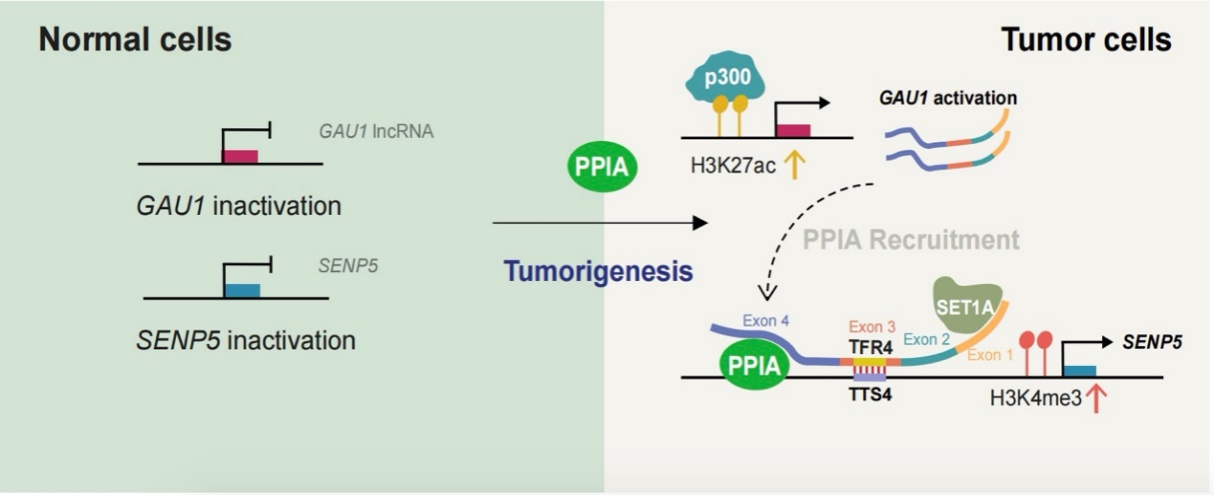
为了研究DNA-lncRNA三链体在肿瘤发生中的作用和调控机制,首先确定了此前课题组发现并命名的促癌lncRNA GAU1作为三链体候选分子。通过预测发现,GAU1在整个基因组中有48,086个三链体潜在形成位点。利用ChIP-qPCR技术,还发现在肿瘤细胞中,GAU1的启动子区域有大量的p300蛋白富集,引起GAU1启动子区域的H3K27乙酰化水平增加,激活了GAU1转录。
接下来,使用ChIRP-MS和RIP-qPCR捕获了与GAU1结合的蛋白,确认PPIA与GAU1直接相互作用。利用Triplex-seq,识别了与GAU1形成DNA-lncRNA三链体的全基因组位点,并通过ChIP-seq分析了PPIA全基因组结合位点。Triplex-seq、ChIP-seq共定位分析和ChIRP-qPCR发现,GAU1和PPIA共同定位在癌基因SENP5启动子上,同时形成了GAU1/SENP5三链体。
为了明确GAU1/SENP5三链体与PPIA在肿瘤发生中的调控机制,利用三链体捕捉技术、EMSA、溶解曲线和圆二色谱法等实验发现,PPIA与GAU1 lncRNA的第四个外显子结合后,锚定GAU1到癌基因SENP5启动子区,随后诱导GAU1的第三个外显子TFR4 RNA片段与SENP5启动子区的TTS4 DNA片段形成SENP5/GAU1 DNA-lncRNA三链体。这种SENP5/GAU1三链体能进一步吸引甲基转移酶SET1A富集到GAU1的第一个外显子,增加H3K4me3激活SENP5转录,显著促进肿瘤细胞生长和转移。该研究还发现,敲除PPIA后,能显著减少SENP5/GAU1 DNA-lncRNA三链体形成,降低SENP5启动子区的H3K4三甲基化富集,并诱导癌基因SENP5转录失活,抑制肿瘤生长和转移,表明PPIA发挥着关键的三链体形成支架蛋白的作用。
综上所述,该研究揭示了肿瘤发生中蛋白引导的DNA-lncRNA三链体形成新机制,为靶向异常的DNA-lncRNA三链体探索潜在肿瘤治疗靶点提供了新认识。
同济大学张晓宇博士、丁天宜博士和博士研究生杨帆、张继星是该文的共同第一作者,同济大学张赫教授为该文唯一通讯作者。同时,作为同济大学对口支援井冈山大学的联合学术成果,该工作还获得了同济大学生命科学与技术学院、同济大学附属东方医院、器官发育与表观遗传江西省重点实验室、井冈山大学附属医院临床医学研究中心、井冈山大学医学部和生命科学学院的大力支持,受到了国家重点研发计划、国家自然科学基金委和上海市科委等项目的资助。
论文链接:https://www.nature.com/articles/s41467-024-53493-x
参考文献:
1. Goni, J.R., X. de la Cruz, and M. Orozco, Triplex-forming oligonucleotide target sequences in the human genome. Nucleic Acids Res, 2004. 32(1): p. 354-60.
2. Xu, Y., et al., Modulation of Hoogsteen dynamics on DNA recognition. Nat Commun, 2018. 9(1): p. 1473.
3. Jalali, S., et al., Genome-Wide Computational Analysis and Validation of Potential Long Noncoding RNA-Mediated DNA-DNA-RNA Triplexes in the Human Genome. Methods Mol Biol, 2021. 2254: p. 61-71.
4. Senturk Cetin, N., et al., Isolation and genome-wide characterization of cellular DNA:RNA triplex structures. Nucleic Acids Res, 2019. 47(5): p. 2306-2321.
5. Leisegang, M.S., et al., RNA-DNA triplexes: molecular mechanisms and functional relevance. Trends Biochem Sci, 2024. 49(6): p. 532-544.